Negli ultimi anni il team di ricerca della Genetica Medica di Siena diretto dalla Professoressa Alessandra Renieri si è focalizzato sullo studio della terapia genica in diverse malattie, fra cui la Sindrome di Rett (RTT). In particolare il team sta lavorando sulla correzione di mutazioni nei geni MeCP2 (c.473C>T (p.(Thr158Met))), FOXG1 (c.688C>T (p.(Arg230Cys))) e CDKL5 (c.190G>T (p.(Glu364*))), che causano rispettivamente la forma classica, la forma congenita e la variante con convulsioni a esordio precoce della malattia. I tre geni hanno un ruolo essenziale per lo sviluppo e il funzionamento del sistema nervoso; quindi se mutati, risulta fondamentale correggere la sequenza in modo preciso senza alterare ulteriormente la loro espressione. Per tale motivo è stata esclusa la tecnica del gene replacement che nella maggior parte dei casi prevede l’aggiunta di una copia addizionale del gene per sopperire alla funzione di quello mutato; questo processo risulta rischioso e potrebbe inficiare ulteriormente sulla sua già alterata espressione. Per tale motivo, il team della prof.ssa Renieri si è focalizzato su una terapia genica basata sul gene editing, che consente di correggere in modo preciso una specifica sequenza all’interno del gene di interesse senza alterarne i meccanismi di regolazione.
L’obiettivo finale degli studi è quello di correggere la mutazione direttamente nei neuroni, il tipo cellulare primariamente coinvolto nella RTT, utilizzando la tecnologia CRISPR/Cas9 e sfruttando uno dei meccanismi di riparazione della cellula, l’Homology-Directed Repair-HDR. Il CRISPR/Cas9 è un sistema di immunità adattiva utilizzato dai batteri per difendersi dai virus e ad oggi è sfruttato dai ricercatori di tutto il mondo per la manipolazione del genoma delle cellule eucariotiche. L’HDR è uno dei meccanismi che le cellule utilizzano per riparare le rotture del DNA a doppio filamento.
La ricerca svolta presso il laboratorio di Genetica Medica ha come vantaggio quello di avere a disposizione un vasto numero di pazienti che vengono presi in carico e seguiti dal team di medici della struttura. Per gli esperimenti di editing sono state utilizzate le cellule dei pazienti ottenute tramite biopsia cutanea, un metodo non invasivo che permette di avere un modello cellulare con la mutazione di interesse. Dalla biopsia si possono isolare i fibroblasti, le cellule cutanee da cui, tramite riprogrammazione genetica, si possono derivare le cellule staminali pluripotenti indotte (induced Pluripotent Stem Cell iPSCs). Le iPSCs sono fondamentali per lo studio di malattie del neuro-sviluppo perché possono essere differenziate in neuroni, permettendo così di studiare le cellule principalmente colpite dalla malattia. Per la correzione delle mutazioni, in collaborazione con l’Istituto per lo Studio, la Prevenzione e la Rete Oncologica (ISPRO), sono stati disegnati e ingegnerizzati due plasmidicheesprimonoicomponenti fondamentali per il gene editing. In particolare, uno dei due plasmidi, il Cas9 vector, esprime l’endonucleasi Cas9, una proteina che funziona come una forbice molecolare, ovvero taglia il DNA nel sito specifico della mutazione e innesca i meccanismi di riparazione e il successivo ripristino della sequenza corretta. Il Cas9 vector codifica anche per due sequenze di auto-inattivazione che hanno lo scopo di limitare l’espressione della proteina all’interno della cellula riducendo il rischio di tagli non specifici sul DNA (off-target) e l’attivazione di meccanismi di difesa all’interno della cellula. Il secondo plasmide, chiamato Targeting vector, codifica per due componenti fondamentali del sistema CRISPR: il primo è l’RNA guida (single guide RNA sgRNA), una sequenza specifica di RNA che riconosce la regione di DNA mutata e dirige sul sito bersaglio la nucleasi Cas9 per l’editing; il secondo è il Donor, una sequenza di DNA che viene usata come stampo dopo il taglio della Cas9 per la sintesi del nuovo filamento con la sequenza corretta. Questo plasmide contiene inoltre un sistema Reporter che permette di individuare e selezionare solo le cellule in cui si esprimono entrambi i plasmidi, composto da due marcatori fluorescenti: mCherry, che si esprime quando il Targeting vector entra nella cellula, che in questo caso apparirà rossa, e GFP (Green Fluorescent Protein), che si esprime solo quando la Cas9 è attiva e in questo caso la cellula è sia rossa che verde. Il sistema basato sull’utilizzo di due plasmidi è stato recentemente brevettato dall’Università di Siena.
La scelta di lavorare con due plasmidi nasce dall’esigenza di utilizzare i virus Adeno-Associati (Adeno Associated Virus – AAVs) come sistema per far arrivare all’interno delle cellule, in vitro e in vivo, i componenti del macchinario di editing. Gli AAVs sono dei virus che non possono contenere DNA di dimensioni elevate, ma rappresentano un sistema virale più sicuro rispetto ad altri sistemi virali utilizzati in terapia genica. Nel nostro caso è stato scelto il sierotipo AAV9 che è in grado di raggiungere il sistema nervoso centrale attraversando la barriera emato-encefalica ed infetta preferenzialmente le cellule neuronali.
La nostra strategia sperimentale prevede la scelta dell’RNA guida specifico per la mutazione, la produzione dei plasmidi di correzione, la validazione della loro efficienza di correzione e il loro incapsidamento nei virus per testare anche in questo caso l’efficienza di correzione nelle cellule del paziente. I primi test di editing su cellule con mutazioni nei geni FOXG1 e MeCP2 hanno dato risultati estremamente promettenti. In primo luogo sono stati trasfettati i fibroblasti dei pazienti; l’attivazione e l’efficienza dei plasmidi sono state validate qualitativamente e quantitativamente utilizzando il microscopio a fluorescenza e la citometria a flusso (Fluorescence-Activated Cell Sorting – FACS). Dopo aver selezionato e recuperato solo i fibroblasti GFP positivi, che quindi esprimono entrambi i plasmidi di correzione, il gene mutato è stato interamente sequenziato attraverso sequenziamento di nuova generazione (Next Generation Sequencing – NGS). Nello specifico, per quanto riguarda la mutazione nel gene MeCP2, c.473C>T (p.(Thr158Met)), sono state analizzate le cellule ottenute da 4 pazienti diversi. I risultati hanno evidenziato un’alta efficienza di HDR, che va da un 20 % ad un 80 %, e una bassa percentuale di inserzioni e delezioni. Vista la percentuale di correzione variabile, è stata analizzata la possibile correlazio-
ne tra l’attività di TP53 e l’efficienza di HDR. TP53 è un soppressore tumorale, anche conosciuto come il guardiano del genoma; esso svolge un ruolo essenziale nella regolazione del ciclo cellulare e, in caso di danni al DNA, induce l’attivazione dei meccanismi di riparazione o la morte della cellula. In letteratura è riportato che il taglio del doppio filamento di DNA causato dalla Cas9 induce un’attivazione di TP53 che andrebbe ad ostacolare il processo di riparazione tramite HDR. Per tale motivo, l’inibizione o la minore funzionalità della proteina potrebbero aumentare l’efficienza di HDR. Nei nostri studi è stato analizzato un polimorfismo funzionale di TP53, c.215C>G (p.(Pro72Arg)), cioè un cambiamento della sequenza di aminoacidi della proteina in cui una Prolina viene sostituita da una Arginina; questa modifica altera la stabilità della proteina e la sua funzione. Tre dei pazienti con mutazione in MeCP2 su cui è stato testato l’editing hanno una sequenza proteica che contiene l’Arginina e in queste pazienti la percentuale di HDR è più alta; al contrario nella quarta paziente la proteina TP53 contiene la Prolina e questa paziente è quella con HDR più basso. Questo risultato suggerisce una possibile correlazione tra stabilità di TP53 e efficienza di HDR; sarà necessario fare ulteriori analisi su un numero maggiore di pazienti al fine di confermare questa ipotesi.
La valutazione dell’efficienza di correzione tramite NGS è stata fatta anche sui fibroblasti mutati in FOXG1 (c.688C>T (p.(Arg230Cys))) trattati con plasmidi di correzione specifici per questa mutazione; in questo caso l’efficienza di correzione è di circa il 22% e la percentuale di inserzioni e delezioni è più alta rispetto a quella ottenuta nelle cellule con mutazione in MeCP2.
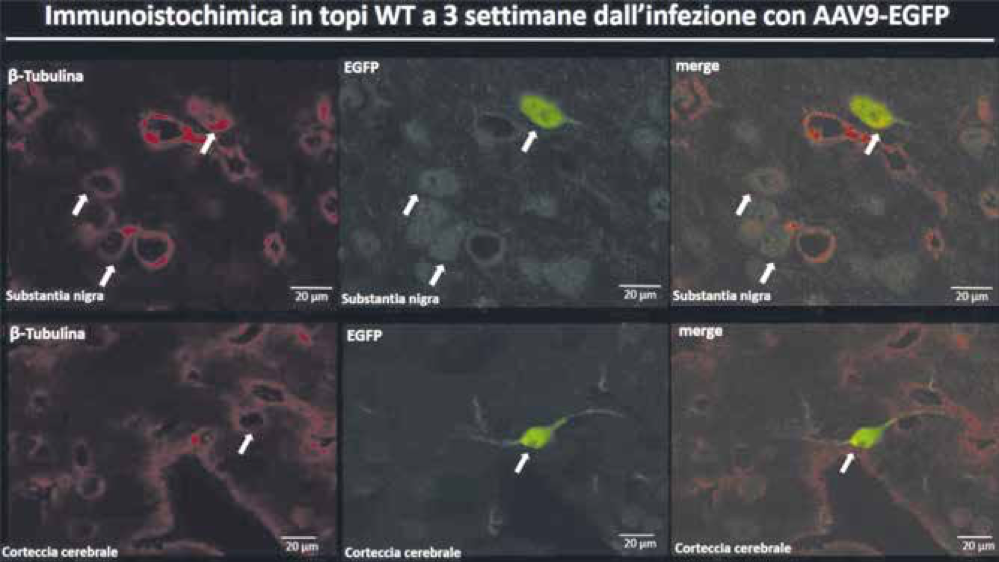
Visti i risultati ottenuti nei fibroblasti, abbiamo testato i nostri plasmidi di correzione nei neuroni differenziati da iPSCs paziente-specifiche. Anche in questo caso sono stati ottenuti incoraggianti risultati di editing; in particolare per quanto riguarda i neuroni MeCP2 l’efficienza di HDR è del 14% con percentuale trascurabile di inserzioni e delezioni; per i neuroni FOXG1 l’efficienza di editing è del 34%, ma in questo caso la percentuale di inserzioni e delezioni è più alta. Questi risultati hanno dimostrato che è possibile fare gene editing nelle cellule del sistema nervoso utilizzando l’HDR e rappresentano un buon punto di partenza per i prossimi esperimenti di editing che prevedono di attuare la stessa strategia utilizzando i virus. A tale scopo, sono stati effettuati dei test di infezione in diversi tipi cellulari. Inizialmente, per ottimizzare i protocolli, sono stati testati due sierotipi diversi, AAV2 e AAV9, che esprimono solamente la GFP.
I risultati hanno evidenziato che gli AAV2 infettano con una buona efficienza cellule poco differenziate come iPSCs e precursori neuronali; al contrario gli AAV9 infettano cellule maggiormente differenziate come neuroni e fibroblasti. Inoltre, esperimenti preliminari in vitro hanno dimostrato che i neuroni infettati con AAV9 che codificano le componenti del sistema di correzione mutazione-specifico, esprimono entrambe le fluorescenze del nostro Reporter (mCherry e GFP), confermando la corretta attivazione del sistema. Il prossimo step sarà quello di validare l’efficienza di HDR in vivo in topi knock-in, cioè animali nei quali è stata inserita la stessa mutazione presente nei pazienti. Grazie alla collaborazione con l’Università di Ferrara, sono stati fatti alcuni test preliminari su topi sani somministrando per via endovenosa AAV9 di controllo che esprimono solo la GFP; a 3 settimane dall’infezione i topi sono stati sacrificati ed è stata fatta un’analisi immunoistochimica su sezioni di varie regioni del cervello, tra cui corteccia cerebrale e substantia nigra. I risultati ottenuti sono riportati nella Fig.1: le sezioni sono state trattate con due anticorpi diversi: l’anti-tubulinaIII-Beta, che colora di rosso i neuroni, e l’anti-GFP per evidenziare ulteriormente la presenza della GFP. Dall’immagine si evince che l’AAV9 è in grado di raggiungere dal sangue periferico il sistema nervoso centrale e rappresenta un buon sistema per veicolare il sistema di correzione direttamente nel cervello.
Per quanto riguarda CDKL5 stiamo completando il disegno dei plasmidi di correzione per la mutazione c.190G>T (p.(Glu364*)); in collaborazione con l’European Molecular Biology Laboratory (EMBL) di Monterotondo è in corso la generazione di un modello murino che presenta la mutazione presente nella paziente e che servirà per gli esprimenti in vivo.

